第二代测序(Next-generation sequencing,NGS)又称为高通量测序,其开创性的引入了可逆终止末端,从而实现边合成边测序,在DNA复制过程中通过捕捉新添加的碱基所携带的特殊标记来确定DNA序列。
二代测序有两个重要特点:1.高通量,二代测序能一次并行对几十、几百万条DNA分子进行测序;2.读长短,测序过程随着读长增长,基因簇复制的协同性降低,会导致测序质量下降,二代测序的读长不超过500bp。因此基因组、宏基因组需要被打断成小片段再测序,测序完毕后再拼接。
总的说来,第一代测序技术的主要特点是测序读长可达1000bp,准确性高达99.999%,但其测序成本高,通量低等方面的缺点,严重影响了其真正大规模的应用。经过不断的技术开发和改进,以Roche公司的454技术、illumina公司的Solexa,Hiseq技术和ABI公司的Solid技术为标记的第二代测序技术诞生了。第二代测序技术大大降低了测序成本的同时,还大幅提高了测序速度,并且保持了高准确性,以前完成一个人类基因组的测序需要3年时间,而使用二代测序技术则仅仅需要1周,但在序列读长方面比起第一代测序技术则要短很多。目前主流的二代测序平台是illumina,下文主要介绍illumina平台的测序原理。
Single-Read测序(Single-read)首先将DNA样本进行片段化处理形成200-500bp的片段,**引物序列连接到DNA片段的一端**,然后末端加上接头,将片段固定在flow cell上生成DNA簇,上机测序单端读取序列。该方式建库简单,操作步骤少,常用于小基因组、转录组、宏基因组测序。
Paired-end文库制备是指在构建待测DNA文库时在**两端的接头上都加上测序引物结合位点**,在第一轮测序完成后,去除第一轮测序的模板链,用对读测序模块(Paired-End Module)引导互补链在原位置再生和扩增,以达到第二轮测序所用的模板量,进行第二轮互补链的合成测序。

第一步: 构建DNA文库
利用超声波把待测的DNA样本打断成小片段,目前除了组装之外和一些其他的特殊要求之外,主要是打断成200-500bp长的序列片段,并在这些小片段的两端添加上不同的接头,构建出单链DNA文库。
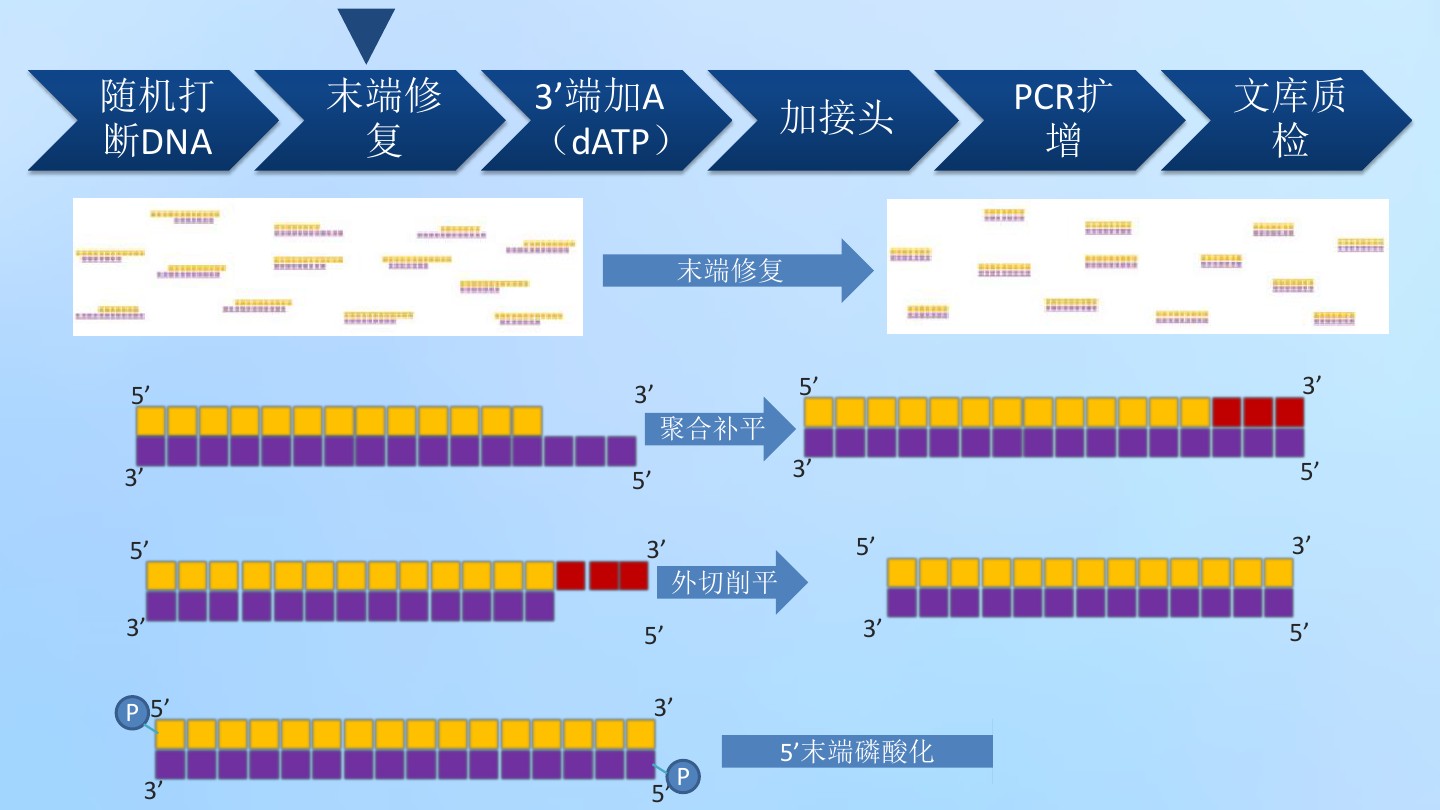
1.末端修饰。打断的片段是随机断裂,其末端可能是不平的。因此,建库第一步是补齐不平的末端。补平之后,需要在3’端加A碱基
.jpg)
2.添加接头。经过末端修饰后的DNA片段3’末端具有突出的A尾,而接头具有突出的T尾,可以使用连接酶将接头添加到DNA片段两端,形成“Y”形接头。
.jpg)
3.PCR扩增。添加了接头的DNA片段,可通过与接头互补的引物来扩增,富集文库
.jpg)
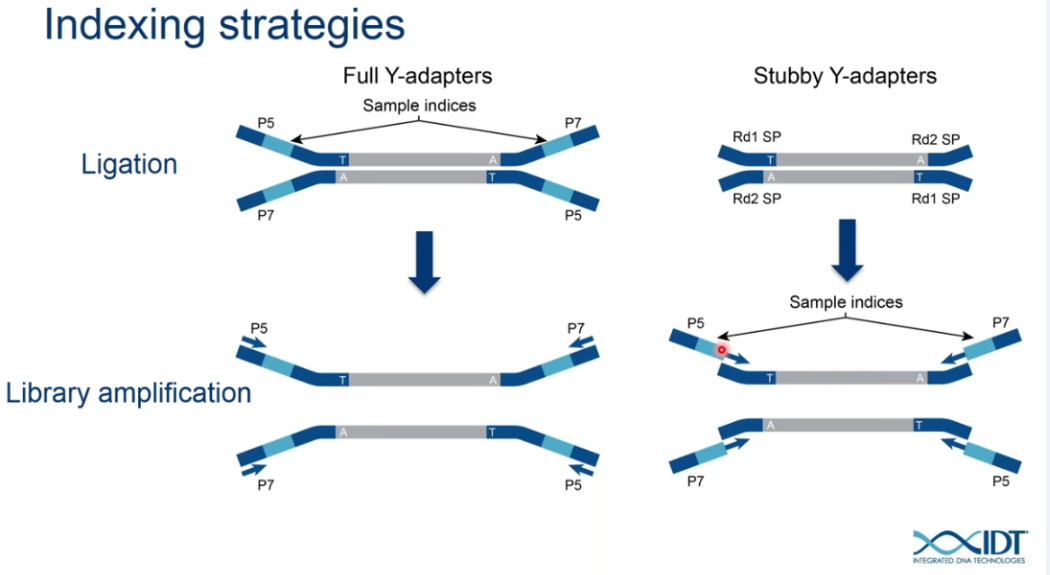
左边的是直接在fragment DNA的两端直接加上full Y-adapter, adapter中已经包括了和P5/P7 oligo互补的序列, index, 以及Read1/Read2的测序引物。
右边的那种是先在fragment DNA的两端加上PE adapter, 然后再引入和P5/P7 oligo互补配对的序列以及index序列。
一句话总结,这两种不同的indexing strategy的差别在于引入index序列的时机和方式不一样。
其实右边的图并不是画的特别形象,具体的的可以参看下面这张图
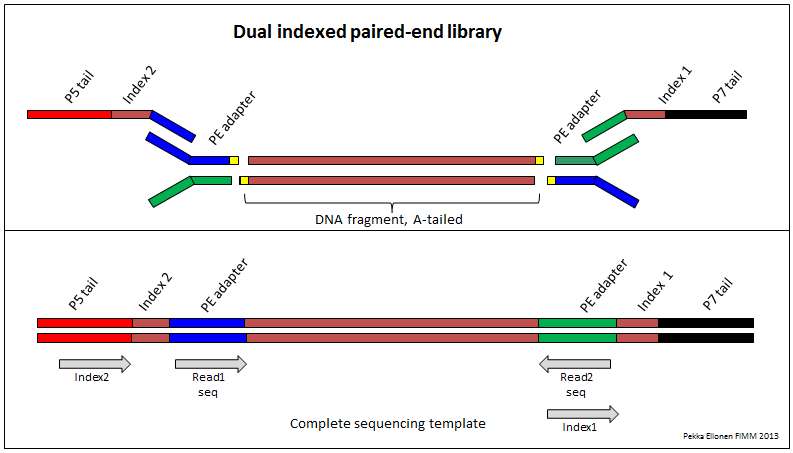
在这里我们能够清楚地看到,这种接头添加过程中,fragment DNA两端是先连上PE adapter, 然后再通过PCR引入的region complementary to P5/P7 sequence, index, and sequencing biding sites.
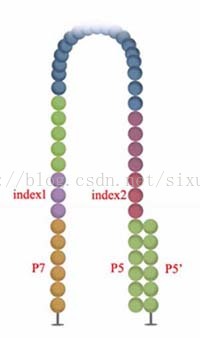
1)P5和P7是不同的,它们分别和flowcell上的接头互补和相同。
2)index1和index2也是不同的,与P5相连的是index2,与P7相连的是index1
关于index,也叫barcodes,因为一个lane可以同时测多个样品,为了避免混淆样品的read products,每种样品的DNA由一种index修饰,这样测序得到的reads都是具有index标记的,在测序结果中,依据之前标签与样品的对应关系,就可以获得对应样品的数据。而这里的index1和index2是为了区分paired-end测序得到的双端reads。
第二步:桥式PCR
Flowcell是用于吸附流动DNA片段的槽道,当文库建好后,这些文库中的DNA在通过flowcell的时候会随机附着在flowcell表面的lane上。每个Flowcell有8个lane,每个lane的表面都附有很多接头,这些接头能和建库过程中加在DNA片段两端的接头相互配对(这就是为什么flowcell能吸附建库后的DNA的原因),并能支持DNA在其表面进行桥式PCR的扩增。
.jpg)
1.Flowcell上随机分布了两种不同的寡核苷酸序列,分别与P5互补(即P5’),与P7一致(即P7)。
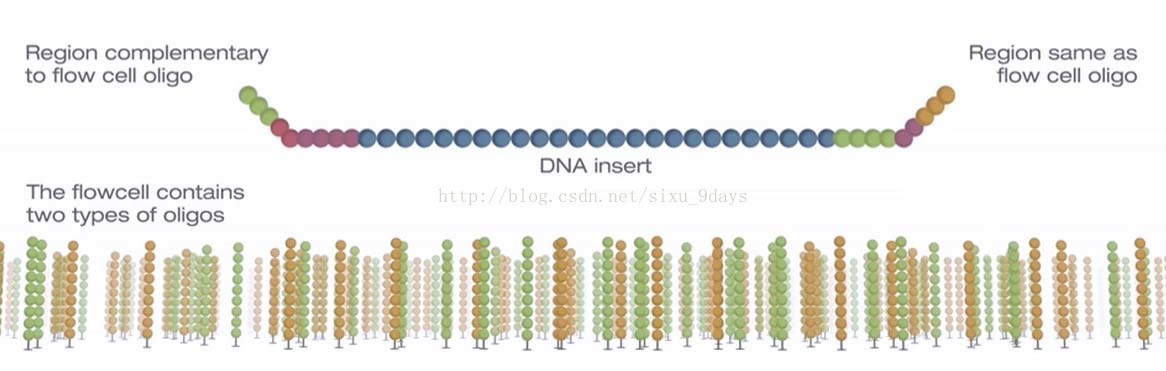
2.待测sequence通过P5与folwcell上的P5’序列杂交互补,以待测sequence为模板进行互补链(即reverse strand)的延伸,互补链的两端为P5’和P7’。

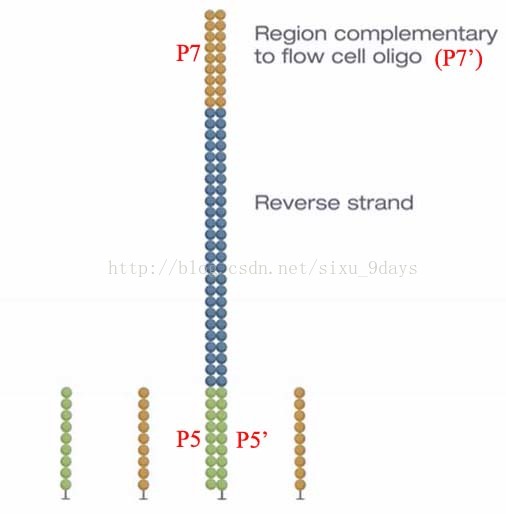
3.通过聚合酶生成杂交片段的互补片段,然后加入NaOH碱溶液后,双链分子变性,原始模板链(左边的链)被流动池中的液体洗去
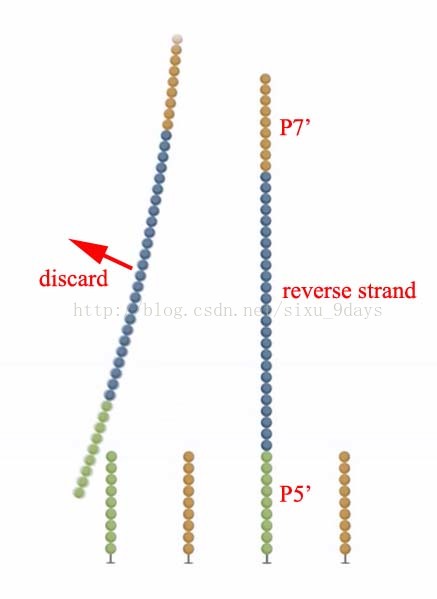
4.加入中性液体用于中和碱溶液,Reverse strand的P7’与Flowcell上的P7杂交互补,进行链的合成,聚合酶参与下,生成互补链,最终形成双链桥,这就是我们所熟知的桥式PCR
5.接下来合成的双链被解链,再分别与Flowcell上的接头杂交互补,延伸,解链,杂交,延伸,解链…如此重复35个循环
6.桥式PCR完成后,使用NAOH将双链解链,并利用甲酰胺基嘧啶糖苷酶(Fpg)对8-氧鸟嘌呤糖苷(8-oxo-G)的选择性切断作用,选择性地将P5’与链的连接切断,留下与Flowcell上P7连接的链,也就是Forward strand。同时游离的3’端被阻断,防止不必要的DNA延伸

第三步:测序
首先,在Flowcell中加入荧光标记的dNTP和酶,由引物起始开始合成子链。但是dNTP存在 3’端叠氮基会阻碍子链延伸,这使得每个循环只能测得一个碱基。合成完一个碱基后, Flowcell 通入液体洗掉多余的dNTP和酶,使用显微镜的激光扫描特征荧光信号。
测序引物(sequencing primer)结合到靠近P5的测序引物结合位点1(sequencing primer binding site 1)上,在系统中加入四种dNTP和DNA聚合酶。这里的dNTP有两个特点:它是有荧光基团标记的,每种碱基标记的荧光基团不一样;它的3’末端连了一个叠氮基,这个叠氮基能够阻断后面的碱基与它相连
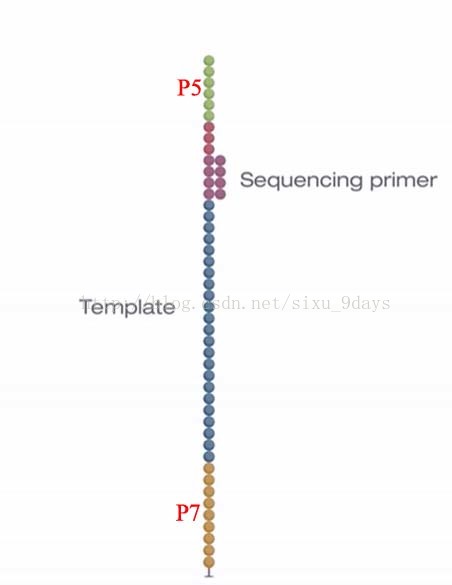
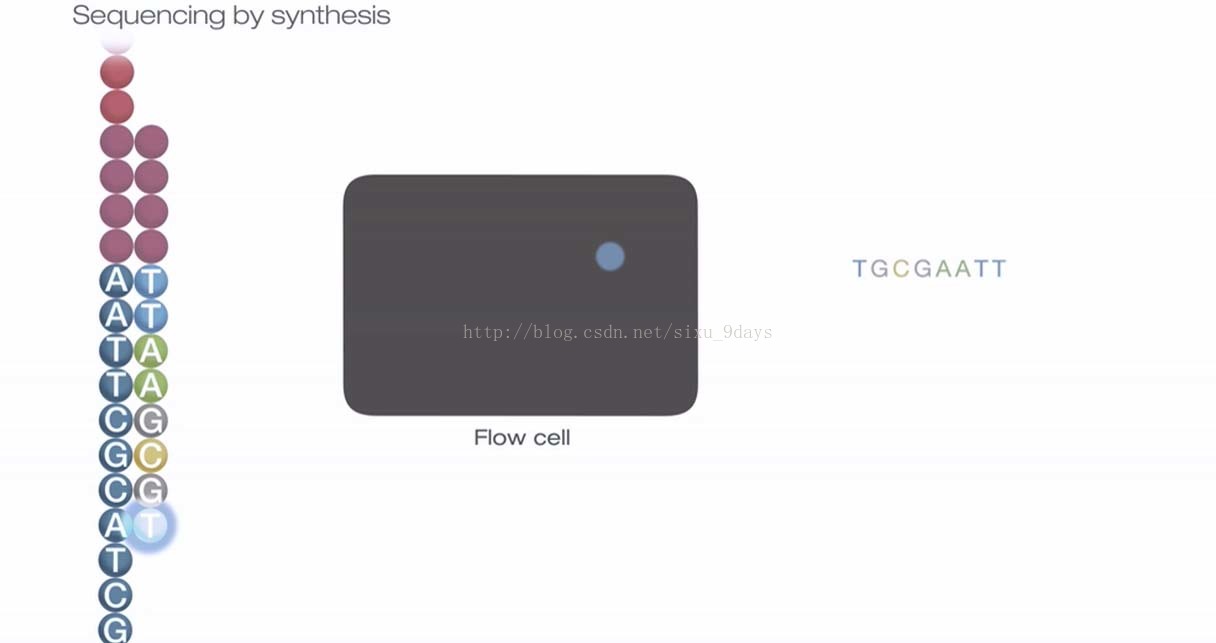
因此在聚合酶的作用下,与Forward strand相应位置碱基配对的dNTP就会结合到新合成的链上,而由于叠氮基的存在,后面的dNTP无法继续连接。这时用水将剩余的dNTP和酶给冲掉,将Flowcell进行扫描,扫描出来的荧光对应的碱基的配对碱基即是该链该位置的碱基。同时在这个Flowcell上有成千上万个cluster也在进行同样的反应,因此一个循环就能同时检测多个样本(这也是高通量的核心所在)。这个循环完成后,加入化学试剂把叠氮基和标记的荧光基团切掉,进行下一个循环(碱基的连接、检测与切除)。如此重复直至所有链的碱基序列被检测出,也就是Forward read 序列。
2.Index测序:所有循环结束后,read products 被洗掉,index1 primer与链上index primer1 结合位点杂交配对,进行index1的合成及检测
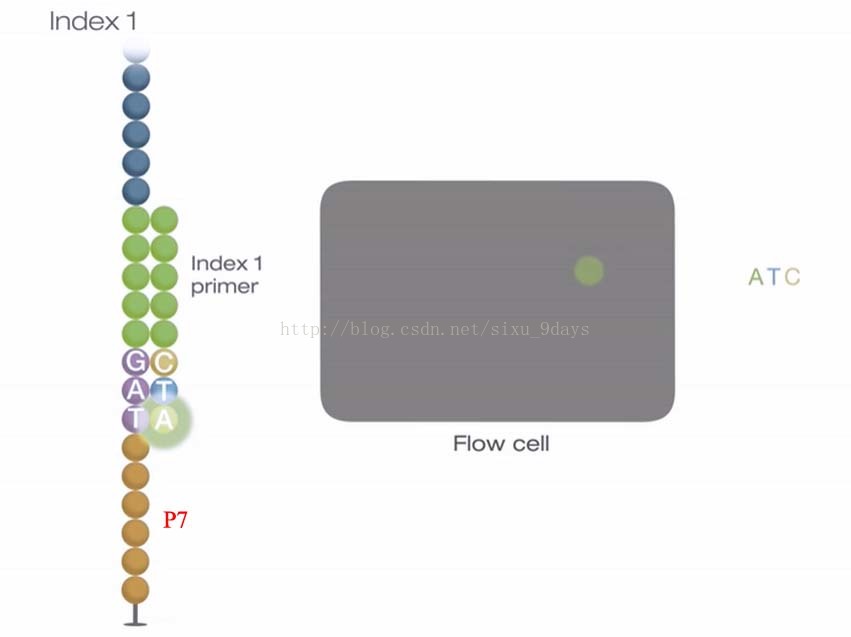
3.Index1测序完成后,洗脱测序产物。此时机器已通过荧光得到了index1的序列

4.Index2测序:Forward strand顶端的P5序列与Flowcell上的P5’杂交配对,进行index2测序。测序完成后洗脱产物
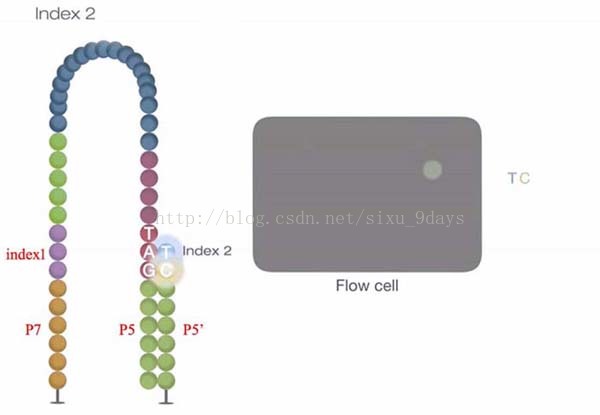
Paried-end sequencing(即对Reverse strand测序)
Paired-end测序已经是现在的主流,它提高了测序长度的同时,又可以为结构变异分析提供新方法。要完成双末端测序,首先要将模板链3’去保护,模板折叠,index片段引入
1.洗脱index2测序产物后,以Flowcell上的P5’为引物,Forward strand为模板进行桥式扩增,得到双链
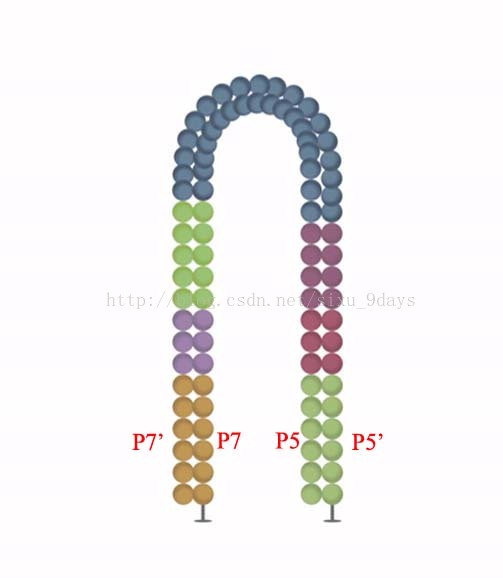
2.NAOH使双链变性为单链,并洗去已经测序完成的Forward strand
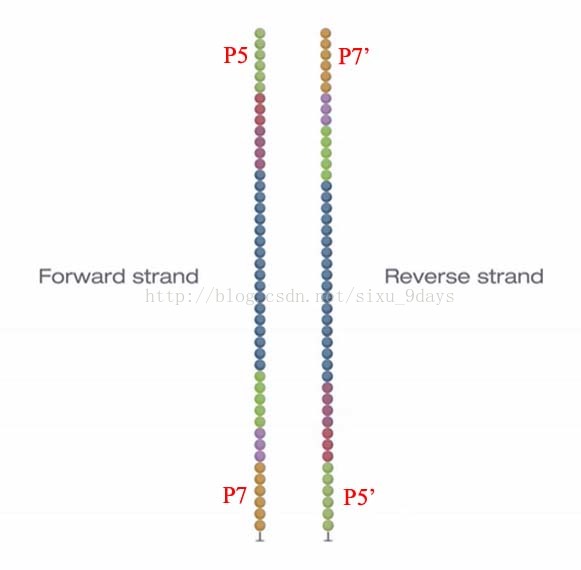

3.类似的,readprimer2结合到靠近P7’的read primer binding site 2开始对Reverse strand的测序。测序完成后即可得到Reverse read序列。

荧光发射波长与信号强度一起决定了碱基的读出,所有的DNA片段的一个碱基会被同时读取。在大规模并行的过程中,机器读取的图像类似下面这样
加入化学试剂将叠氮基团与荧光基团切除,然后 Flowcell 再通入荧光标记的dNTP和酶,由引物起始开始合成一个碱基。不断重复这个过程,完成第一次读取
引用
1.测序之前篇: NGS测序中,接头是如何添加上的,以及如何去接头
3.二代测序文库构建

